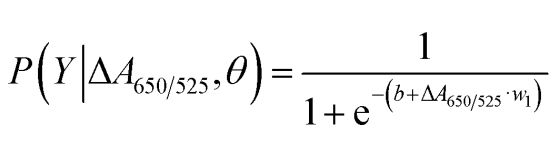Tidig upptäckt av cancer baserad på flytande biopsi är en ny inriktning för cancerdetektering och diagnos som föreslagits av US National Cancer Institute under senare år, med syftet att upptäcka tidig cancer eller till och med precancerösa lesioner. Det har använts i stor utsträckning som en ny biomarkör för tidig diagnos av olika maligniteter, inklusive lungcancer, gastrointestinala tumörer, gliom och gynekologiska tumörer.
Framväxten av plattformar för att identifiera biomarkörer i metyleringslandskapet (Methylscape) har potential att avsevärt förbättra befintlig tidig screening för cancer, vilket placerar patienter i det tidigaste behandlingsbara stadiet.
Nyligen har forskare utvecklat en enkel och direkt sensorplattform för detektion av metyleringslandskap baserad på cysteamin-dekorerade guldnanopartiklar (Cyst/AuNP) i kombination med en smartphone-baserad biosensor som möjliggör snabb tidig screening av ett brett spektrum av tumörer. Tidig screening för leukemi kan utföras inom 15 minuter efter DNA-extraktion från ett blodprov, med en noggrannhet på 90,0 %. Artikeltiteln är Snabb detektion av cancer-DNA i mänskligt blod med hjälp av cysteamin-kapslade AuNP och en maskininlärningsaktiverad smartphone.
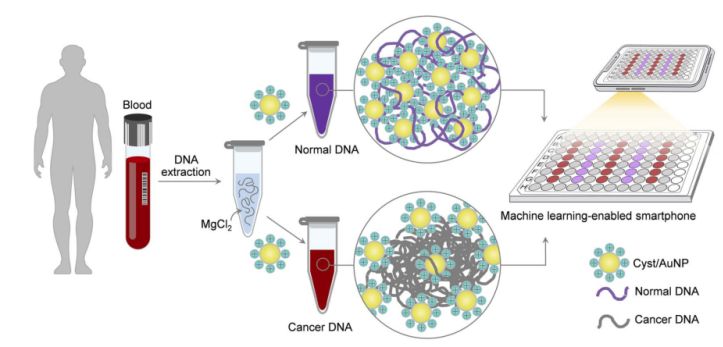
Figur 1. En enkel och snabb sensorplattform för cancerscreening via Cyst/AuNP-komponenter kan åstadkommas i två enkla steg.
Detta visas i figur 1. Först användes en vattenlösning för att lösa upp DNA-fragmenten. Cyst/AuNP tillsattes sedan till den blandade lösningen. Normalt och malignt DNA har olika metyleringsegenskaper, vilket resulterar i DNA-fragment med olika självorganiseringsmönster. Normalt DNA aggregerar löst och aggregerar så småningom Cyst/AuNP, vilket resulterar i Cyst/AuNP:s rödskiftade natur, så att en färgförändring från rött till lila kan observeras med blotta ögat. Däremot leder den unika metyleringsprofilen för cancer-DNA till produktion av större kluster av DNA-fragment.
Bilder av 96-brunnsplattor togs med en smartphonekamera. Cancer-DNA mättes med en smartphone utrustad med maskininlärning jämfört med spektroskopibaserade metoder.
Cancerscreening i riktiga blodprover
För att utöka användbarheten av sensorplattformen använde forskarna en sensor som framgångsrikt kunde skilja mellan normalt och canceröst DNA i riktiga blodprover. Metyleringsmönster vid CpG-ställen reglerar epigenetiskt genuttryck. I nästan alla cancertyper har förändringar i DNA-metylering och därmed i uttrycket av gener som främjar tumörbildning observerats alternera.
Som en modell för andra cancerformer associerade med DNA-metylering använde forskarna blodprover från leukemipatienter och friska kontroller för att undersöka metyleringslandskapets effektivitet för att differentiera leukemiska cancerformer. Denna biomarkör för metyleringslandskapet överträffar inte bara befintliga snabba leukemiscreeningmetoder, utan visar också möjligheten att utvidga det till tidig upptäckt av ett brett spektrum av cancerformer med hjälp av denna enkla och okomplicerade analys.
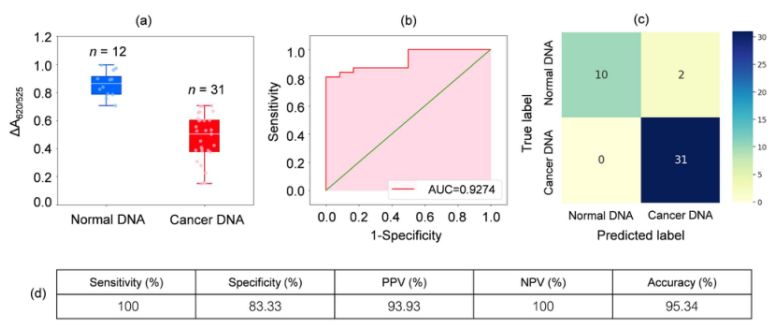
DNA från blodprover från 31 leukemipatienter och 12 friska individer analyserades. Som visas i boxplottet i figur 2a var den relativa absorbansen hos cancerproverna (ΔA650/525) lägre än den för DNA från normala prover. Detta berodde främst på den ökade hydrofobiciteten som ledde till tät aggregation av cancer-DNA, vilket förhindrade aggregation av Cyst/AuNP. Som ett resultat dispergerades dessa nanopartiklar fullständigt i de yttre skikten av canceraggregaten, vilket resulterade i en annan dispersion av Cyst/AuNP adsorberade på normala och cancer-DNA-aggregat. ROC-kurvor genererades sedan genom att variera tröskelvärdet från ett minimivärde på ΔA650/525 till ett maximivärde.
Figur 2.(a) Relativa absorbansvärden för cyst-/AuNP-lösningar som visar närvaron av normalt (blått) och cancer- (rött) DNA under optimerade förhållanden.
(DA650/525) av boxdiagram; (b) ROC-analys och utvärdering av diagnostiska tester. (c) Förvirringsmatris för diagnos av normala patienter och cancerpatienter. (d) Sensitivitet, specificitet, positivt prediktivt värde (PPV), negativt prediktivt värde (NPV) och noggrannhet hos den utvecklade metoden.
Som visas i figur 2b uppvisade arean under ROC-kurvan (AUC = 0,9274) som erhölls för den utvecklade sensorn hög känslighet och specificitet. Som framgår av boxplottet är den lägsta punkten som representerar den normala DNA-gruppen inte väl separerad från den högsta punkten som representerar cancer-DNA-gruppen; därför användes logistisk regression för att skilja mellan de normala och cancergrupperna. Givet en uppsättning oberoende variabler uppskattar den sannolikheten för att en händelse ska inträffa, såsom en cancer- eller normalgrupp. Den beroende variabeln varierar mellan 0 och 1. Resultatet är därför en sannolikhet. Vi bestämde sannolikheten för canceridentifiering (P) baserat på ΔA650/525 enligt följande.
där b=5,3533,w1=-6,965. För urvalsklassificering indikerar en sannolikhet på mindre än 0,5 ett normalt urval, medan en sannolikhet på 0,5 eller högre indikerar ett cancerurval. Figur 2c visar den förvirringsmatris som genererats från "leave-it-alone"-korsvalideringen, vilken användes för att validera klassificeringsmetodens stabilitet. Figur 2d sammanfattar den diagnostiska testutvärderingen av metoden, inklusive sensitivitet, specificitet, positivt prediktivt värde (PPV) och negativt prediktivt värde (NPV).
Smartphone-baserade biosensorer
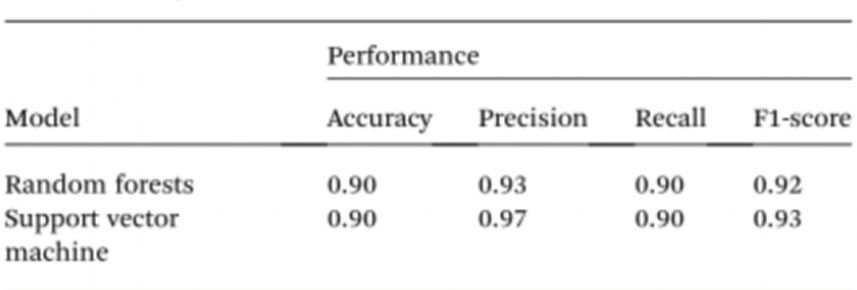
För att ytterligare förenkla provtestning utan användning av spektrofotometrar använde forskarna artificiell intelligens (AI) för att tolka lösningens färg och skilja mellan normala och cancersjuka individer. Med detta i åtanke användes datorseende för att översätta färgen på Cyst/AuNPs-lösningen till normalt DNA (lila) eller cancersjukt DNA (rött) med hjälp av bilder av 96-brunnsplattor tagna genom en mobiltelefonkamera. Artificiell intelligens kan minska kostnaderna och förbättra tillgängligheten vid tolkning av färgen på nanopartikellösningar, och utan användning av optisk hårdvara eller smartphonetillbehör. Slutligen tränades två maskininlärningsmodeller, inklusive Random Forest (RF) och Support Vector Machine (SVM), för att konstruera modellerna. Både RF- och SVM-modellerna klassificerade korrekt proverna som positiva och negativa med en noggrannhet på 90,0 %. Detta tyder på att användningen av artificiell intelligens inom mobiltelefonbaserad biosensing är fullt möjlig.
Figur 3. (a) Målklass för lösningen registrerad under beredningen av provet för bildtagningssteget. (b) Exempelbild tagen under bildtagningssteget. (c) Färgintensitet hos cysta/AuNP-lösningen i varje brunn i 96-brunnsplattan extraherad från bilden (b).
Med hjälp av cystor/aunopartiklar har forskare framgångsrikt utvecklat en enkel sensorplattform för metyleringsdetektering och en sensor som kan skilja normalt DNA från cancer-DNA när riktiga blodprover används för leukemiscreening. Den utvecklade sensorn visade att DNA extraherat från riktiga blodprover snabbt och kostnadseffektivt kunde detektera små mängder cancer-DNA (3 nM) hos leukemipatienter på 15 minuter, och visade en noggrannhet på 95,3 %. För att ytterligare förenkla provtestning genom att eliminera behovet av en spektrofotometer användes maskininlärning för att tolka lösningens färg och skilja mellan normala och cancerösa individer med hjälp av ett mobiltelefonfotografi, och en noggrannhet kunde också uppnås på 90,0 %.
Referens: DOI: 10.1039/d2ra05725e
Publiceringstid: 18 februari 2023
 中文网站
中文网站